Nowa rola rdzeniowych białek spliceosomu (białek Sm) w metaboliznie mRNA
Kontrolowana ekspresja określonych genów w komórce jest podstawą funkcjonowania wszystkich organizmów żywych. Znaczącą rolę w tym procesie odgrywają zdarzenia potranskrypcyjne zachodzące w cytoplazmie. Wiele etapów potranskrypcyjnej obróbki i dojrzewania mRNA zachodzi na terenie wyspecjalizowanych ciał cytoplazmatycznych. Te konserwowane ewolucyjnie mikrodomeny stanowią miejsce regulacji czasu translacji oraz degradacji mRNA, przez co funkcjonują jako centrum koordynacji równowagi między tymi procesami. Dostępna literatura opisuje głównie ciała cytoplazmatyczne w komórkach zwierzęcych. U roślin ich funkcje skład molekularny pozostają znacznie mniej poznane. Jak dotąd, w komórkach roślinnych nie opisano występowania kanonicznych białek Sm w ciałach cytoplazmatycznych bogatych w mRNA.
Białka Sm to rodzina konserwowanych ewolucyjnie małych białek wiążących RNA, występujących u wszystkich żywych organizmów. W komórkach eukariotycznych uczestniczą one w splicingu pre-mRNA. Najnowsze badania wskazują jednak, że Sm mogą regulować los mRNA również na późniejszych etapach dojrzewania. Jak wykazały nasze ostatnie badania na terenie cytoplazmy biorą one w zupełnie odmiennymi procesami związanymi z metabolizmem mRNA (Hyjek i wsp. 2019; Hyjek-Składanowska i wsp. 2020).
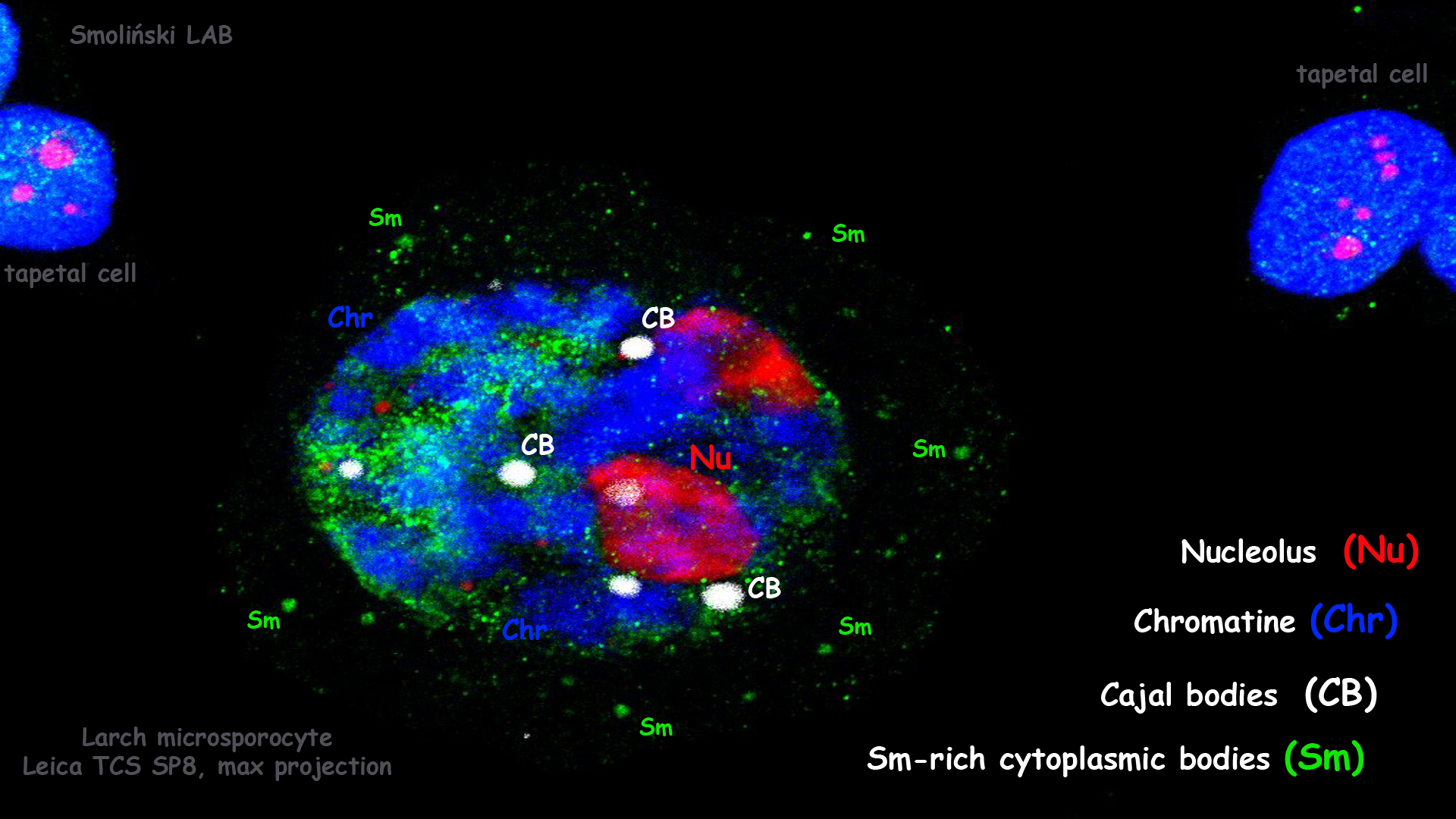 Celem tego projektu jest weryfikacja hipotezy, że kanoniczne białka Sm są częścią cytoplazmatycznego kompleksu mRNP i pełnią istotną rolę w post-transkrypcyjnej regulacji ekspresji genów u roślin. Projekt realizujemy w oparciu o metody bioobrazowania RNA i białek w komórce jak i metody biologii molekularnej takie jak NGS i RIP-seq.
Celem tego projektu jest weryfikacja hipotezy, że kanoniczne białka Sm są częścią cytoplazmatycznego kompleksu mRNP i pełnią istotną rolę w post-transkrypcyjnej regulacji ekspresji genów u roślin. Projekt realizujemy w oparciu o metody bioobrazowania RNA i białek w komórce jak i metody biologii molekularnej takie jak NGS i RIP-seq.

 ul. Lwowska 1, 87-100 Toruń
ul. Lwowska 1, 87-100 Toruń